Veröffentlicht am von Illa
Im Jahr 2003 entstand im Hamburger „Bernhard-Nocht-Institut für Tropenmedizin“ (BNI) der PCR-Test auf das Virus SARS-CoV, das das Schwere akute respiratorische Syndrom SARS verursachen soll. Entwickelt wurde er damals von Christian Drosten und seinem Kollegen Stephan Günther, auch Olfert Landt war mit TIB Molbiol schon dabei. [1] Seitdem haben Drosten und Landt diverse PCR-Protokolle bzw. ‑Kits geliefert einschließlich der Zutaten für den aktuellen „Drosten-Test“ schlechthin. Dieses PCR-Protokoll wurde am 13./14. Januar 2020 von der WHO veröffentlicht und die Publikation in einer Fachzeitschrift erfolgte wenig später: „Corman VM, Landt O, Kaiser M, et al. Detection of 2019 novel coronavirus (2019 nCoV) by real time RT PCR. Euro Surveill 2020; 25: 2000045“.
Erschienen ist die Arbeit am 23. Januar 2020 in Eurosurveillance [2], nachdem sie am 21. eingereicht und am Folgetag angenommen worden war – ein absoluter Rekord für einen als „Research“ deklarierten Artikel [3]. Für die Autoren war es wichtig, in dieser und nicht erst eine Woche später in der nächsten Ausgabe des Journals zu erscheinen und den Wettlauf um die Erstpublikation als Voraussetzung für viele Zitationen zu gewinnen, die wiederum in der akademischen Welt mitbestimmend für Sein und Nichtsein sind. Inzwischen ist die stattliche Anzahl von über 7000 Zitationen zusammengekommen [4] – allerdings haben dazu auch Kritiker des PCR-Protokolls beigetragen wie die Autoren des „Corman-Drosten Review Reports“ [5], die aufgrund schwerer Mängel von Eurosurveillance gefordert hatten, den Artikel von Corman et al. zurückzuziehen. Hier geht es um die Frage, ob das PCR-Protokoll für SARS-CoV‑1 von 2003 schon die gleichen Fehler aufwies wie sein aktueller Nachfolger für SARS-CoV‑2. Als Richtschnur dient dazu die Fehleranalyse des „Corman-Drosten Review Reports“.
Immer zu wenig Zielgene, falsche Anordnung: „was den Test … ungeeignet macht“
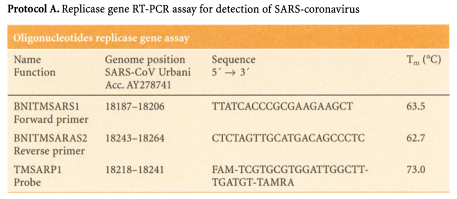
Drosten und weitere 25 Autoren (darunter die Niederländer Ron Fouchier und Albert Osterhaus sowie Herbert Schmitz vom BNI) publizierten 2003 im „New England Journal of Medicine“ (NEJM) die Arbeit „Identification of a Novel Coronavirus in Patients with Severe Acute Respiratory Syndrome“ [1]. Darin enthalten ist die Abbildung 1 mit dem Protokoll 6 für die PCR – genauer: die meist verwendete Realtime-PCR, deren Verlauf „in Echtzeit“ verfolgt werden kann. Dies sind die Angaben für die Testung (zwei Primer, eine Sonde) auf ein Zielgen, das sich auf der ORF1b-Region (Replikase-Gen RdRp) befindet und einen Abschnitt (Amplikon) von 77 Nukleotiden (bp) umfasst.
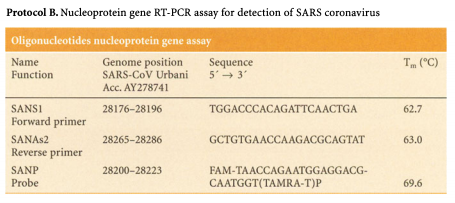
2004 publizierte Drosten als alleiniger Autor den Beitrag „Detection of SARS-Coronavirus in the LightCycler by S‘-Nuclease Real-Time RT-PCR“ für ein Handbuch zur „Rapid Cycle Real-Time PCR“. [6] Darin war zusätzlich zum ORF1b-Protokoll ein weiteres zum N‑Gen von 128 Nukleotiden enthalten, das aber wohl erst zur Verfügung stand, als SARS schon wieder vorbei war. Drosten schrieb zu seinen Testanleitungen in diesem Buch:
„Sie können verwendet werden, um eine gültige Bestätigung von SARS in Übereinstimmung mit den von der WHO während der SARS-Epidemie 2003 herausgegebenen Richtlinien zu erhalten […]. Positive Befunde in einem der beiden Assays können durch den anderen querbestätigt werden. […]
Laboratorien, die RT-PCR zur Bestätigung von SARS einsetzen wollen, sollten sich darüber im Klaren sein, dass von der WHO vorgeschrieben wird, positive Befunde durch Wiederholung des Tests zu bestätigen
– aus einem anderen klinischen Probentyp des selben Patienten (z. B. Atemwegsprobe und Stuhl), oder
– aus dem gleichen Probentyp, der zu einem anderen Zeitpunkt entnommen wurde, oder
– aus derselben Probe mit einem Assay, der auf eine andere Genom-Region abzielt.
Idealerweise sollten immer zwei oder mehr Tests, die auf unterschiedliche Genom-Regionen abzielen, angewendet werden.“ [6]
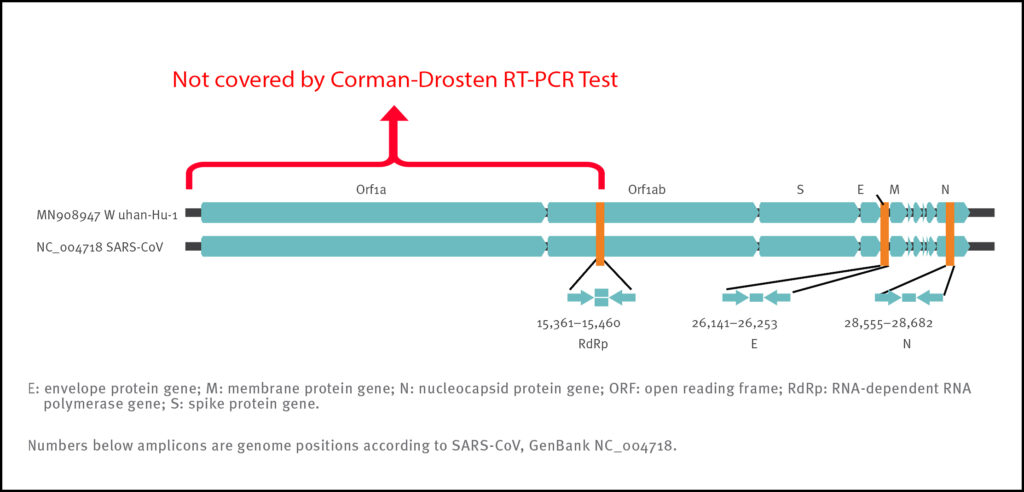
Das Genom der SARS-Coronaviren ist ca. 30.000 Nukleotide lang. Der von den Primern begrenzte Abschnitt des Gens für ORF-1b hat die Position 18.187–18.264, der für das N‑Gen 28.176–28.286 – gemeinsam belegen sie den Bereich von 18.187 bis 28.286 [6] und damit ca. 10.000 Nukleotide, umfassen also nur ein Drittel des gesamten Genoms von SARS-CoV‑1. Damit kann bestenfalls angenommen werden, dass dieses Stück in der entnommenen Probe intakt vorhanden war – für den großen Rest gibt der Test keinerlei Informationen. Ein solches Bruchstück würde jedoch das gleiche Signal geben wie ein vollständig vorhandenes Genom und damit ist die positive Reaktion beider Zielgene kein Beleg für das Vorhandensein des intakten Virusgenoms geschweige denn die Fähigkeit des Virus zur Replikation als Voraussetzung für dessen Infektiosität. Das war auch ein Kritikpunkt am „Drosten-Test“:
„Selbst wenn wir also drei positive Signale (d.h. die drei Primerpaare ergeben 3 verschiedene Amplifikationsprodukte) in einer Probe erhalten, beweist dies nicht das Vorhandensein eines Virus. Ein besseres Primerdesign würde terminale Primer an beiden Enden des viralen Genoms haben. Dies liegt daran, dass das gesamte virale Genom abgedeckt wäre und drei positive Signale besser zwischen einem vollständigen (und damit potentiell infektiösen) Virus und fragmentierten viralen Genomen (ohne infektiöse Potenz) unterscheiden können.“ [5]
Bei den drei 2020 in Eurosurveillance genannten Zielgenen (RdRp‑, E‑, N‑Gen) wird etwa die Hälfte des Genoms nicht von den Tests erfasst, wie in der Graphik veranschaulicht wird. Weggelassen wurde aber schon im Januar 2020 das N‑Gen, wodurch sich der erfasste Bereich von 15.361 bis 26.253 erstreckt, also – wie bei SARS-CoV‑1 – auf ca. 10.000 Nukleotide und damit nur auf ein Drittel des gesamten Genoms. Seit Frühjahr 2020 ist es sogar üblich, nur auf das E‑Gen zu testen [7], also weniger als 1% des Genoms, um dann zu behaupten, man habe das Virus nachgewiesen und daraus eine Infektiosität mitsamt diversen „Maßnahmen“ plus „Impfung“ abzuleiten.
Diese Abbildung für die Kritik am „Drosten-Test“ läßt sich auf das PCR-Protokoll von 2003 übertragen und damit gilt nicht nur für SARS-CoV‑2, sondern auch für SARS-CoV‑1: „Der Test kann nicht zwischen dem ganzen Virus und viralen Fragmenten unterscheiden. Daher kann der Test nicht als Diagnostikum für intakte (infektiöse) Viren verwendet werden, was den Test als spezifisches Diagnosewerkzeug zur Identifizierung des SARS-CoV-2-Virus und zur Ableitung von Rückschlüssen auf das Vorliegen einer Infektion ungeeignet macht.“ [5]
Immer zu viele Zyklen, kein Cutoff: „so dass der Test … ungeeignet ist“
Die Zyklenzahl ist ein weiterer wichtiger Parameter der PCR und zugleich eine mögliche – und auch praktizierte – Stellschraube, um nach Bedarf eine große Anzahl positiver Testergebnisse zu generieren.
„Im Falle des Virusnachweises werden bei >35 Zyklen nur Signale erkannt, die nicht mit infektiösen Viren korrelieren, wie sie durch Isolierung in Zellkulturen bestimmt werden […]; wird jemand durch PCR positiv getestet, wenn ein Schwellenwert von 35 Zyklen oder höher verwendet wird (wie es in den meisten Labors in Europa und den USA der Fall ist), beträgt die Wahrscheinlichkeit, dass die betreffende Person tatsächlich infiziert ist, weniger als 3 %, die Wahrscheinlichkeit, dass es sich bei dem Ergebnis um ein falsches Positiv handelt, 97 % […].
PCR-Daten, die nach einem Ct-Wert von 35 Zyklen als positiv bewertet werden, sind völlig unzuverlässig. […]
Zwischen 30 und 35 gibt es eine Grauzone, in der ein positiver Test nicht mit Sicherheit festgestellt werden kann. Dieser Bereich sollte ausgeschlossen werden. Natürlich könnte man 45 PCR-Zyklen durchführen, wie im Corman-Drosten WHO-Protokoll empfohlen […], aber dann muss man auch einen vernünftigen Ct-Wert definieren (der 30 nicht überschreiten sollte). Ein Analyseergebnis mit einem Ct-Wert von 45 ist jedoch wissenschaftlich und diagnostisch absolut bedeutungslos […] Es ist ein erheblicher Fehler, dass in dem Corman-Drosten-Papier der maximale Ct-Wert, bei dem eine Probe eindeutig als positives oder negatives Testergebnis gewertet werden kann, nicht erwähnt wird.“ [5]
Die für den „Drosten-Test“ angegebene Zyklenzahl von 45 ist absurd hoch und die 2003 von 40 nur unwesentlich besser, zumal in beiden Fällen kein Ct-Wert angegeben war, also kein Schwellenwert, bis zu dem eine Probe als positiv gilt. Sowohl für SARS-CoV‑1 als auch für SARS-CoV‑2 gilt daher: „Ein schwerwiegender Fehler ist das Fehlen eines Ct-Werts, bei dem eine Probe als positiv und negativ gilt. Dieser Ct-Wert findet sich auch nicht in den Folgeanträgen, so dass der Test als spezifisches Diagnoseinstrument zum Nachweis des SARS-CoV-2-Virus ungeeignet ist.“ [5]
Die Primer aus den Protokollen von 2003 und 2020 wurden von Olfert Landt durch TIB Molbiol kommerzialisiert. Zu seinen Produkten existieren Manuals mit technischen Daten und Gebrauchsanweisungen. Während im Manual von 2003 bei einer Zyklenzahl von 40 kein Ct-Wert enthalten war [8], war 2020 bei einer Zyklenzahl von 45 immerhin ein Ct-Wert von 39 angegeben [9]. Landt war diese Anforderung inzwischen also klar geworden, sie fand sich aber nicht in den von ihm mitverfaßten Artikeln für die WHO und Eurosurveillance wieder und angesichts eines Ct-Werts, „der 30 nicht überschreiten sollte“ ist 39 offenbar auch noch immer viel zu hoch.
MERS-Intermezzo
Es gab also sowohl 2003/4 als auch 2020 das Problem der Zielgene und der Zyklenzahl, doch waren diese Fehler bei SARS‑2 sogar noch schwerwiegender als bei SARS‑1. Zwischen diesen beiden Tests lag 2012 der auf MERS-CoV als Verursacher des „Middle East Respiratory Syndrome“, der ebenfalls von Corman et al. bei Eurosurveillance publiziert wurde: „Detection of a novel human coronavirus by real-time reverse-transcription polymerase chain reaction“ [10] Der Artikel wurde am 27. September 2012 eingereicht und noch am selben Tag veröffentlicht, was auch für die Rubrik Rapid Communications abnorm schnell ist [3] – und möglicherweise dadurch befördert wurde, dass Drosten seit mindestens 2008 Mitherausgeber (Associate Editor) von Eurosurveillance war. [11] Auch bei diesem Artikel ist er – damals noch Professor in Bonn – letztgenannter und damit verantwortlicher Autor. Zwischen ihm und seinem Doktoranden Corman sind u.a. Fouchier und Osterhaus sowie Landt anzutreffen, dessen Firma extra bedacht wurde: „Oligonukleotide können ab Lager bei Tib-Molbiol, Berlin bestellt werden (www.tib-molbiol.de)“. [10]
In diesem Artikel werden die PCR-Anleitungen für zwei Zielgene beschrieben. Das E‑Gen wird als Screening Assay und das ORF1b-Gen als Bestätigungstest eingesetzt, das N‑Gen wird zwar kurz erwähnt, aber wieder verworfen. Bis auf einen ORF-1b-Primer (36%) haben alle Primer und auch die Sonden einen GC-Gehalt zwischen 40 und 60% (s.u. „GC-Gehalt (prozentualer Gehalt des Basenpaars Guanin/Cytosin“). Es wird eine sehr hohe Primerkonzentration von 400 nmol verwendet, (s.u. „Primerkonzentration“), die Zyklenzahl ist mit 45 angegeben.
Ein Nachtrags-Artikel erschien am 6. Dezember 2012, nachdem er am Vortag eingereicht worden war wiederum unter Rapid Communications. Darin war das Zielgen für den Bestätigungstest ORF-1a statt ORF-1b und alle Primer lagen gut im GC-Bereich von 40–60%. Corman und Drosten bilden wieder Beginn und Ende der Autorenliste, dazwischen ist Fouchier verschwunden, während u.a. Andreas Nitsche dazugekommen ist, [12] der bei TIB Molbiol war, bevor er 2002 an das Robert-Koch-Institut wechselte [13] Geblieben waren 45 Zyklen ohne Ct-Wert und 400 nmol Primerkonzentration aus dem ersten MERS-Artikel.
Mit diesen Parametern war die Empfindlichkeit des Tests auf Kosten der Genauigkeit im Vergleich zu SARS erhöht worden. Drosten drückte den mangelnden diagnostische Wert eines solchen „viel bringt viel“-Ansatzes zwei Jahre später so aus:
„Als in Dschidda Ende März diesen Jahres aber plötzlich eine ganze Reihe von Mers-Fällen auftauchten, entschieden die dortigen Ärzte, alle Patienten und das komplette Krankenhauspersonal auf den Erreger zu testen. Und dazu wählten sie eine hochempfindliche Methode aus, die Polymerase-Kettenreaktion (PCR). […]
Ja, aber die Methode ist so empfindlich, dass sie ein einzelnes Erbmolekül dieses Virus nachweisen kann. Wenn ein solcher Erreger zum Beispiel bei einer Krankenschwester mal eben einen Tag lang über die Nasenschleimhaut huscht, ohne dass sie erkrankt oder sonst irgend etwas davon bemerkt, dann ist sie plötzlich ein Mers-Fall. Wo zuvor Todkranke gemeldet wurden, sind nun plötzlich milde Fälle und Menschen, die eigentlich kerngesund sind, in der Meldestatistik enthalten. Auch so ließe sich die Explosion der Fallzahlen in Saudi-Arabien erklären.“ [14]
Für Landt, der an beiden MERS-Publikationen beteiligt war, wurde kein Interessenkonflikt deklariert. Seit 2014 gibt es bei TIB Molbiol MERS-Kits für das E- und das Orf1a-Gen mit einer Anleitung für 45 Zyklen und der Bewertung „positiv“ bei <39 Zyklen [15], ein Verfahren, das er auch bei seinen C19-Kits beibehalten hat. Im Jahr 2020 verkündete Landt im Zusammenhang mit C19, die Hälfte der Test-Positiven sei nicht infektiös [16] womit er die Kritikpunkte an dem von ihm mitverfaßten Testprotokoll und den von ihm vertriebenen Kits wenigstens ansatzweise bestätigte.
Mehrere Verschlimmbesserungen
Weitere Kritikpunkte trafen allerdings auf das SARS-Protokoll von 2003 noch nicht zu, damals waren diese Standards offenbar bekannt und wurden eingehalten.
- Primerkonzentration
Im PCR-Protokoll wird in der Rezeptur auch die Konzentration der Primer in Nanomol (nmol) angegeben, es geht also darum, wie viele Primer für die Reaktion zur Verfügung stehen. Beim „Drosten-Test“ ist sie ungewöhnlich hoch.
„Es gibt keinen bestimmten Grund, diese extrem hohen Konzentrationen von Primern in diesem Protokoll zu verwenden. Die beschriebenen Konzentrationen führen zu erhöhten unspezifischen Bindungen und PCR-Produktamplifikationen, wodurch der Test als spezifisches Diagnoseinstrument zum Nachweis des SARS-CoV-2-Virus ungeeignet ist.“ [5]
Als übliche Primerkonzentration wird ein Wert von 100 bis 200 nmol angegeben [5] und dieser Bereich wurde 2003 eingehalten. 2020 aber waren sowohl für das RdRp- als auch das N‑Gen für je einen der Primer Konzentrationen von 800 nmol angegeben (in der Kombination 800 und 600 nmol für das Primerpaar), beim E‑Gen waren es 400 nmol für beide Primer. [1]
- Wobbly positions
In zwei Primern und einer Sonde des „Drosten-Tests“ auf das RdRp-Gen (das spezifisch für SARS-Cov‑2 sein soll) gibt es „wobbly positions“, was sich mit Wackelpositionen übersetzen läßt. Gemeint ist damit ein nicht definiertes Nukleotid in der Sequenz, für das es jeweils zwei Möglichkeiten gibt: z.B. steht W für Adenin oder Thymin. Daraus ergibt sich keine konkrete Primersequenz, sondern mehrere Varianten.
„Diese hohe Anzahl von Varianten ist nicht nur ungewöhnlich, sondern auch sehr verwirrend für die Labors. […] Die Design-Variationen führen unweigerlich zu Ergebnissen, die nicht einmal mit SARS CoV‑2 in Verbindung stehen. […] Diese unspezifizierten Positionen hätten eindeutig gestaltet werden müssen. […]
Sechs nicht spezifizierte wobbly positions werden eine enorme Variabilität in die realen Laborimplementierungen dieses Tests einführen; die verwirrende unspezifische Beschreibung im Corman-Drosten-Papier ist nicht als Standard-Betriebsprotokoll geeignet, was den Test als spezifisches Diagnoseinstrument zur Identifizierung des SARS-CoV-2-Virus ungeeignet macht.“ [5]
Im Artikel „So veränderte der Drosten-Test den Verlauf der Corona-Pandemie ‚Der Corman-Drosten-Test war eine Meisterleistung‘“ wird der Einsatz von „wobbly positions“ (hier als „degenerierte Primer“ bezeichnet) hochgelobt:
„Echte Virusproben existieren zu diesem Zeitpunkt nur in China. Und niemand weiß, wie repräsentativ die erste Genomsequenz für das neue Coronavirus wirklich ist.
Denn die Erbinformation von Viren ein und derselben Art kann sich an einzelnen Stellen, bei einzelnen Buchstaben im Genom unterscheiden. Corman riskiert also, dass sein mühsam entwickelter PCR-Test Viren übersieht, weil die Primer nicht genau genug zu den Sequenzen der Virusvarianten passen, die tatsächlich in der Mehrzahl grassieren.
Um trotzdem möglichst viele solcher Varianten abzudecken und aufzuspüren, greift der Virologe zu einem klassischen Kniff im Primerdesign: Er lässt an bestimmten Stellen seiner Primer-Sequenzen offen, welcher konkrete Buchstabe dort stehen soll. Fachleute nennen solche PCR-Primer degenerierte Primer. Ausgesuchte Stellen in der Primer-Sequenz der PCR-Reagenzien dürfen sowohl mit der einen als auch mit der anderen Base besetzt sein. Das soll dem PCR-Test etwas mehr Spielraum verschaffen und die Chance erhöhen, die Viren auch dann noch nachzuweisen, wenn sie nicht haargenau passen. Das Vorgehen ist weltweit üblich, zumal in der Frühphase einer Erkrankungswelle mit einem neuen Erreger.“ [17]
Bemerkenswert ist allerdings, dass bei einer Auflistung der sieben frühen PCR-Protokolle aus der WHO-Liste (Januar-Februar 2020) mit insgesamt 17 Primerpaaren/Sonden nur zwei (die Charité bei RdRp und die Universität Hongkong bei ORF1b-nsp14) wobbly positionsaufwiesen [18] und bei SARS und MERS kamen Drosten und Kollegen offenbar ohne diesen „klassischen Trick“ aus.
- Schmelztemperatur (Tm)
Für die Tm des Primerpaares gibt es einen maximaler Differenzwert, damit beide Primer unter gleichen Bedingungen an die Zielsequenz binden oder sich von ihr ablösen. Dieser Standard ist in einem 1998 von Drosten und seinem Doktorvater Kurt Roth angemeldeten (aber nicht erteilten) Patent genau erklärt: „Das für die PCR eingesetzte Primer-Paar weist vorzugsweise eine gleiche Schmelztemperatur (errechnet über die ‚nearest neighbour‘-Methode) oder eine um höchstens 2,5°C unterschiedliche Schmelztemperatur auf. […] Die Schmelztemperatur der Primer liegt dabei geeigneterweise im Bereich von 55°C bis 65°C, vorzugsweise im Bereich von 58°C bis 62°C und insbesondere bei 59°C bis 60°C.“ [19] Drosten hielt diese Standards bei seiner PCR für SARS auch vorbildlich ein.
Beim „Drosten-Test“ 2020 aber wurde auch das über Bord geworfen:
„Die Annealing-Temperatur (Tm) ist ein entscheidender Faktor für die Bestimmung der Spezifität/Genauigkeit des qPCR-Verfahrens und wesentlich für die Bewertung der Genauigkeit von qPCR-Protokollen. Best-Practice-Empfehlung: Beide Primer (vorwärts und rückwärts) sollten einen annähernd gleichen Wert haben, vorzugsweise den gleichen Wert.
[…] Eine maximale Tm-Differenz von 2° C innerhalb der Primerpaare wurde als akzeptabel angesehen. Beim Testen der in der Corman-Drosten-Arbeit angegebenen Primer-Paare haben wir für Primer-Paar1 (RdRp_SARSr_F und RdRp_SARSr_R) eine Differenz von 10° C in Bezug auf die Annealing-Temperatur Tm festgestellt. Dies ist ein sehr schwerer Fehler und macht das Protokoll als spezifisches Diagnosewerkzeug unbrauchbar.“ [5]
- GC-Gehalt (prozentualer Gehalt des Basenpaars Guanin/Cytosin):
Der GC-Gehalt meint den relativen Anteil der Nukleotide Guanin und Cytosin im Verhältnis zur Gesamtheit der vier Nukleotide, aus denen die DNA besteht. Je höher der GC-Gehalt ist, desto stärker ist die Bindung; er hat Einfluß auf die Tm.
„Für eine effiziente und spezifische Amplifikation sollte der GC-Gehalt der Primer mindestens 40 % und maximal 60 % der Amplifikation entsprechen. […]. Zwei Primer (RdRp_SARSr_F und RdRp_SARSr_R) haben ungewöhnliche und sehr niedrige GC-Werte von 28 %-31 % für alle möglichen Varianten von Wobble-Basen, während der Primer E_Sarbeco_F einen GC-Wert von 34,6 % aufweist […].
Es ist zu beachten, dass der GC-Gehalt aufgrund der drei Wasserstoffbrückenbindungen bei der Basenpaarung die Bindung an sein spezifisches Ziel weitgehend bestimmt. Je niedriger also der GC-Gehalt des Primers ist, desto geringer ist seine Bindungsfähigkeit an seine spezifische Zielgensequenz (d. h. das zu detektierende Gen). […]
Wenn der Tm-Wert sehr niedrig ist, wie bei allen Wobbly-Varianten der RdRp-Reverse-Primer beobachtet, können die Primer unspezifisch an mehrere Targets binden, was die Spezifität verringert und potenziell falsch-positive Ergebnisse erhöht.“ [5]
Der GC-Gehalt war 2003 im Standard-Bereich und 2020 gleich bei zwei von drei Zielgenen gab es Werte darunter. Bei MERS hatte es schon einen solchen „Ausreißer“ gegeben, doch damals wurde er immerhin nachträglich korrigiert.
Summa summarum
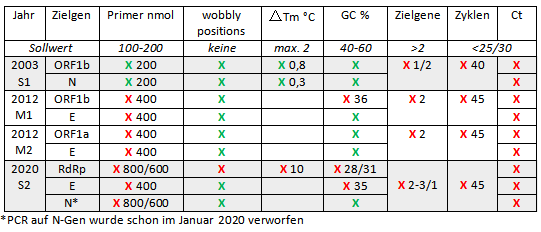
Dies ist eine tabellarische Übersicht über einige Parameter der PCR-Protokolle für SARS1 (2003/4) und C19 (2020) sowie beide Protokolle für MERS (2012). Je grüner desto besser – aber offensichtlich kommt im Laufe der Zeit immer mehr Rot dazu.
Die beiden erstgenannten Mängel (zu wenige Zielgene, zu viele Zyklen bei fehlendem Cutoff) waren schon 2003 vorhanden. Weitere Parameter wie die Konzentration oder die Schmelztemperatur der Primer waren damals zwar standardgemäß, aber das ändert nichts an den zentralen Problemen. Damit gilt schon für SARS-CoV‑1: Mit dieser PCR konnte kein korrekter Virusnachweis erbracht werden, sie war dafür methodisch nicht geeignet.
Damals gab es aber immerhin keine Testung von Gesunden („Asymptomatischen“), es gab also noch den Abgleich mit Symptomen, wenn sie teilweise auch unspezifisch (Fieber, Husten etc.) waren und es gab die Anforderung einer epidemiologischen Verbindung. Diese SARS-Definition [20] grenzte die Zielgruppe so stark ein, dass die extrem empfindliche Methode PCR nach diesem Protokoll eigentlich bei sehr vielen Proben positiv sein müßte. War sie aber nicht.
Dazu sagte Herbert Schmitz vom BNI, der damalige Chef von Drosten und Günther sowie Mitautor des NEJM-Artikels von 2003: „In Kanada haben die Ärzte das Virus tatsächlich nur in 40 Prozent der Fälle gefunden. Jetzt fragt man sich, was bei dem Rest ist. In Hongkong grassiert beispielsweise derzeit eine Influenza, die genau dieselben Symptome hervorruft. Das wird nicht richtig auseinander gehalten. […] Alle Zahlen sind sehr wackelig und müssten dringend bereinigt werden. Ein Beispiel: In Deutschland wurden bisher sieben SARS-Fälle gemeldet. Das Corona-Virus haben wir aber nur in drei von ihnen nachweisen können.“ [21] Was auch weniger als die Hälfte ist. Diese Zahlen führten damals zu Schlagzeilen wie „Ursache von SARS umstritten – Leiter eines kanadischen Labors ist nicht davon überzeugt, dass das Coronavirus SARS verursacht“. [22] Und auch Schmitz fiel auf: „Da geht irgendwas nicht zusammen.“ [21]
Dann war SARS plötzlich vorbei, die relativ enge Definition mit dem untergeordnete Stellenwert von Tests wirkte selbst-limitierend. [20] Das Problem ruhte erstmal, Drosten und Günther wurden mit Bundesverdienstkreuzen geehrt und machten Karriere. Zur Jahreswende 2019/2020 arbeitete Drosten mit Corman und Landt dann wieder an einem ganz ähnlichen PCR-Protokoll wie damals und baute eine Reihe Fehler ein, die bei SARS noch nicht gemacht worden waren. Fehler, die im Lauf der Zeit immer weiter zunehmen, entstehen jedoch nicht aus Unwissen oder mangelndem Können. Sie werden bewusst gemacht und wenn sie alle dazu führen, dass mehr positive Ergebnisse entstehen ohne Rücksicht darauf, ob sie richtig- oder falsch-positiv sind, besteht darin das Ziel des Pfuschs. Unangenehme Schlagzeilen wie 2003 wurden mit diesen absurden Zahlen vermieden, das PCR-Protokoll öffnete die Schleusen und die Deiche wurden unterhöhlt, als die Falldefinition allein an das Ergebnis der PCR geknüpft wurde und zudem noch die Massentests an „Asymptomatischen“ begannen. Darauf kann nichts anderes folgen als die Überschwemmung und „Land unter“.
Wer unbedingt auf C19 testen will, hätte Alternativen gehabt. Eine nannte die Humanbiologin Ulrike Kämmerer in ihrem Gutachten für das Verfahren am Weimarer Familiengericht, das für sie und einige weitere an diesem Gerichtsverfahren Beteiligten Hausdurchsuchungen zur Folge hatte [23]:
„Generell kann eine RT-qPCR schon aufgrund der methodischen Vorgänge keine intakten, vernehmungsfähigen (infektiösen) Viren nachweisen, nicht einmal das komplette intakte Virusgenom, sondern ausschließlich Nukleinsäure des gesuchten Abschnitts. Es ist generell möglich, bei gut eingestellten und korrekt durchgeführten PCR-Tests durch Validierung mit einer parallel durchgeführten Virusanzucht in Zellkultur einen Grenzwert (CT) zu definieren, ab dem ein positives PCR-Signal nicht mehr mit vernehmungsfähigen Viren korreliert. Diese ist in der Überwachung von Blutprodukten seit Jahren gut geübte Routine.
Diese stringente Validierung erlaubt dann – solange das Testsystem NICHT verändert wird – als Surrogatmarker eine Abschätzung der Viruslast und damit der möglichen Infektiosität der getesteten Probe, nie allerdings jedoch den definitiven Nachweis. Sobald eine Komponente am PCR-Testsystem (seien es Chemikalien, Plastikwaren, Enzyme, Protokollabläufe oder Maschinen) in einem der angewendeten Schritte verändert wird, muss zwingend das System wieder neun kalibriert werden.
Aus allen bisher publizierten Informationen […] kann davon ausgegangen werden, dass jeder CT-Wert über 35 nicht mehr mit einer Anzüchtbarkeit infektiöser Viren einhergeht und damit der absolute Grenzwert für die Entscheidung „positiv“ ist, auch unabhängig vom verwendeten Testsystem. Der CT-Bereich 25–35 ist testabhängig möglicherweise noch valide als Surrogatmarker für „positiv im Sinne einer potentiell für eine Infektiosität ausreichenden Viruslast“ zu bewerten, wenn er, wie beschrieben, durch adäquate Validierung im durchführenden Labor mit einer Virusanzucht verglichen wurde.
CT≤ 25 : positiver Genomnachweis hohe mRNA Last in der Probe
CT 26–35 : nur positiv, wenn mit Virusanzucht abgeglichen
CT > 35 : negativ“ [24]
Wäre auf diese Weise getestet worden, wäre uns allen viel erspart geblieben. Oder andersherum: Nur mit dem auf eine maximale Anzahl Positive – wie falsch sie auch sein mögen – ausgerichteten PCR-Protokoll war es möglich, die Voraussetzungen für die „Maßnahmen“ und letztlich für die „Impfungen“ zu schaffen.
– Hervorhebungen in blau von mir –
mehr dazu: